[转载]二代测序技术
高通量测序技术是对传统测序一次革命性的改变,一次对几十万到几百万条DNA分子进行序列测定,因此在有些文献中称其为下一代测序技术 (next generation sequencing)足见其划时代的改变,同时高通量测序使得对一个物种的转录组和基因组进行细致全貌的分析成为可能,所以又被称为深度测序(deep sequencing)。
自从2005年454 Life Sciences公司(2007年该公司被Roche正式收购)推出了 454 FLX焦磷酸测序平台(454 FLX pyrosequencing platform)以来,曾推出过3730xl DNA测序仪(3730xl DNA Analyzer) 的Applied BioSystem(ABI)这家一直占据着测序市场最大份额的公司的领先地位就开始动摇了,因为他们的拳头产品毛细管阵列电泳测序仪系列 (series capillary array electrophoresis sequencing machines)遇到了两个强有力的竞争对 手,一个就是罗氏公司(Roche)的454 测序仪(Roch GS FLX sequencer),,另一个就是2006年美国Illumina公司推出的Solexa基因组分析平台(Genome Analyzer platform),为此,2007年ABI公司推出了自主研发的SOLiD 测序仪(ABI SOLiD sequencer)。这三个测序平台即为目前高通量测序平台的代表。(见表一)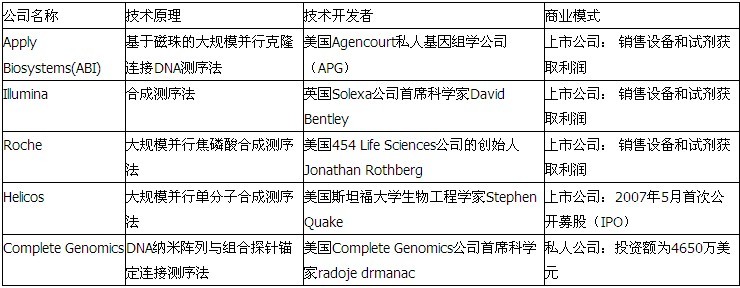
表一:主流测序平台一览
这些平台共同的特点是极高的测序通量,相对于传统测序的96道毛细管测序,高通量测序一次实验可以读取40万到 400万条序列。读取长度根据平台不同从25bp到450bp,不同的测序平台在一次实验中,可以读取1G到14G不等的碱基数,这样庞大的测序能力是传 统测序仪所不能比拟的。尽管如此,在这项新的划时代的测序技术刚出现的时候,科学界对这项新技术却并不热衷。许多习惯用桑格技术的科学家怀疑新技术的准确 度、阅读能力、成本消费、实用性。代理Sanger型测序硬件的经销商害怕其投资失败而首先提出了这些怀疑。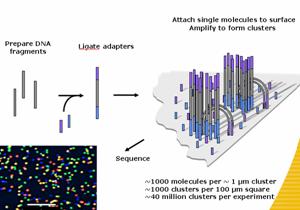
然而大多数人却忽略了一个事实,即桑格技术的普及最初也遇到同样的阻碍。桑格技术刚开发出来时,阅读能力很难超过 25bp,即使在Fred Sanger双脱氧终止法发明后也只达到80bp,如今却达到了750bp;而新发展的合成测序技术,应用焦磷酸测序方法,其阅读能力最初只有 100bp,推向市场16个月后增加至250bp,随着技术的不断完善,目前已达到了400bp,很快就接近桑格技术目前的水平。除了阅读能力外,能否以 有限的成本用一台仪器产生足够数量的序列标记也是另一个需要改善的重要问题。这个问题已经被Roche公司解决了,应用他们的系统,仅花费阅读35bp或 者更小片段的成本就能产生比35bp多10倍的序列标记。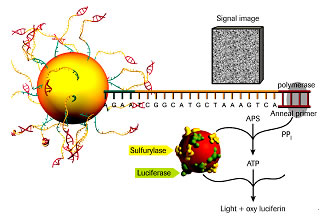
一、高通量测序的应用
高通量测序可以帮助研究者跨过文库构建这一实验步骤,避免了亚克隆过程中引入的偏差。 依靠后期强大的生物信息学分析能力,对照一个参比基因组(reference genome)高通量测序技术可以非常轻松完成基因组重测序(re-sequence),2007年van Orsouw等人结合改进的AFLP 技术和454 测序技术对玉米基因组进行了重测序,该重测序实验发现的超过75%的SNP位点能够用SNPWave技术验证,提供了一条对复杂基因组特别是含有高度重复 序列的植物基因组进行多态性分析的技术路线。2008年Hillier对线虫CB4858 品系进行Solexa重测序,寻找线虫基因组中的SNP位点和单位点的缺失或扩增。但是也应该看到,由于高通量测序读取长度的限制,使其在对未知基因组进 行从头测序(novo sequencing)的应用受到限制,这部分工作仍然需要传统测序(读取长度达到850 碱基)的协助。但是这并不影响高通量测序技术在全基因组mRNA表达谱,microRNA表达谱,ChIP-chip以及DNA甲基化等方面的应用。
2008年Mortazavi等人对小鼠的大脑、肝脏和骨骼肌进行了RNA 深度测序,这项工作展示了深度测序在转录组研究上的两大进展,表达计数和序列分析。对测得的每条序列进行计数获得每个特定转录本的表达量,是一种数码化的 表达谱检测,能检测到丰度非常低的转录本。分析测得的序列,有大于90%的数据显示落在已知的外显子中,而那些在已知序列之外的信息通过数据分析展示的是 从未被报道过的RNA剪切形式,3’端非翻译区,变动的启动子区域以及潜在的小RNA 前体,发现至少有3500个基因拥有不止一种剪切形式。而这些信息无论使用芯片技术还是SAGE文库测序都是无法被发现的。
高通量测序另一个被广泛应用的领域是小分子RNA或非编码RNA(ncRNA)研究。测序方法能轻易的解决芯片技术在检测小分子时遇到的技术难 题(短序列,高度同源), 而且小分子RNA的短序列正好配合了高通量测序的长度,使得数据“不浪费”,同时测序方法还能在实验中发现新的小分子RNA。在衣藻、斑马鱼、果蝇、线 虫、人和黑猩猩中都已经成功地找到了新的小分子RNA。在线虫中获得了40 万个序列,通过分析发现了18个新的小RNA分子和一类全新的小分子RNA。
在DNA—蛋白质相互作用的研究上,染色质免疫沉淀—深度测序(ChIP-seq)实验也展示了其非常大的潜力。染色质免疫沉淀以后的DNA 直接进行测序,对比ref seq可以直接获得蛋白与DNA结合的位点信息,相比ChIP-chip,ChIP-seq可以检测更小的结合区段、未知的结合位点、结合位点内的突变情 况和蛋白亲合力较低的区段。

二、高通量测序的前景
目前,大多分析家都无法相信新一代测序技术能完全取代目前的芯片测序技术。不过,有些分析家也的确认为芯片测序技术正面临着挑战,他们认为到了2012年新一代的测序技术将会带来高达2。15亿美元的产值。
2006年,整个芯片测序市场大概价值8亿美元,其中65%的市场份额都是有关基因表达谱分析产品的,剩下35%的市场份额则由基因型分析芯片 占据。不过美国哈佛大学(Harvard University)遗传学教授George Church认为,这部分市场也会受到新一代测序技术的冲击。重测序芯片(resequencing arrays)、单核苷酸多态性分析芯片以及基因拷贝数目变异分析芯(copy number variant array)市场也会受到影响。也有分析家不赞同这个观点,他们认为即使新一代测序技术很便宜,还是有不少人会选择传统的测序仪的。
新一代测序技术相对传统芯片测序技术的优势,最终还得依靠广告和市场营销手段的推广才能获得大众的认可。去年夏天,由 Frost & Sullivan公司对学术科研机构和私人研究团体进行的一项调查研究结果表明,在实际应用领域,例如进行表达谱分析时,人们还 是倾向于选择传统的芯片产品,而并非青睐新一代的测序产品。
新一代测序仪推广困难可能由其价格昂贵导致。平均采购一台新一代测序仪大约要花费50万美元,除非该实验室测序的工作量非常大,否则是不会考虑 购买的。即使像Polonator这样的新一代测序仪也需要花费15万美元左右,这笔费用对于一个小实验室来说是无法承受的。这时,只需要150美元一块 的芯片就非常有竞争力了。以基因芯片产品享誉业界的美国Affymetrix公司市场部副总裁Jay Kaufman认为,新一代测序技术对于芯片市场来说的确会带来一定的冲击,不过要完全取代表达谱分析芯片还需要一定的时间。
但是,基因芯片也有其自身的缺点,就在于它是一个“封闭系统”,它只能检测人们已知序列的特征(或有限的变异)。而高通量测序的强项, 就在于它是一个“开放系统”, 它的发现能力和寻找新的信息的能力,从本质上高于芯片技术。 研究者可以充分享受这两个平台的比较优势,在获取新信息的基础上,利用芯片的强项, 即对已知信息的高通量、低成本(相对)的检测能力, 对大量样品进行快速检测,短时间内获得有大量有效的数据。
作为两个高通量的基因组学研究技术,在应用的某些方面存在重叠和竞争,但是在更多方面是优势互补,两种方法联合使用,将解决以前的单种技术难以解决的问题。
三、结语
新一代测序已显示出巨大的潜力。也正是因为科学的不断进步,在给测序技术提出新要求的时候,也给这项技术带来了新的增长点:
2008年4月Helico BioScience公司的Timothy等人在Science上报道了他们开发的真正的单分子测序技术,也被称为第三代测序技术,并利用该技术对一个 M13病毒基因组进行重测序。这项技术之所以被称为真正的单分子测序,是因为它完全跨过了上述3种高通量测序依赖的基于PCR扩增的信号放大过程,真正达 到了读取单个荧光分子的能力,向1000美元测定一个人类基因组的目标迈出了一大步。